波江野 洋はえの ひろし
数理生物学
研究テーマ
- 免疫からの攻撃を回避するがんの進化と免疫療法の数理モデル解析
- 肺がん投薬治療における薬剤耐性を回避する最適投薬戦略探索
- クローナル造血の数理モデル研究
- 腫瘍内不均一性がもたらす放射線照射耐性獲得機序の数理モデル解析
経歴
2005年早稲田大学教育学部理学科卒業。2010年九州大学理学府生物科学専攻数理生物学教室(巌佐庸教授)にて博士(理学)を取得。大学院時代、2008年から2009年の間Memorial Sloan-Kettering Cancer CenterのComputational Biology部門(Prof. Franziska Michor)にてVisiting PhD Studentを経験。2010年から2012年までDana-Farber Cancer InstituteのBiostatistics and Computational Biology部門(Prof. Franziska Michor)にてResearch Fellow。2013年より九州大学理学研究院(巌佐庸教授)にて助教、2018年に国立がん研究センター先端医療開発センター(小林進分野長)にて博士研究員、2019年より東京大学大学院新領域創成科学研究科(鈴木穣教授)にて特任准教授。2022年より東京理科大学生命医科学研究所准教授。
がんが正常細胞から発生する過程や薬剤耐性を獲得する過程をヒト体内での細胞の進化過程として捉え、確率モデルや微分方程式系を用いて解析を行っています。計測技術とコンピュータ技術の発展とともに、構築した数理モデルの検証が可能になってきており、より現実に即した数理研究が可能になってきています。がんにも多種多様な病態があり、それぞれのシステムを数理モデルで表現しカタログ化することを目指しています。
大腸がんが免疫から逃れる機序の解明 (Kawazu et al. 2021 Gastroenterology, 日本経済新聞電子版に掲載)

マイクロサテライト不安定性大腸がんは、一定程度で免疫チェックポイント阻害剤の有効性が確認されていますが、必ずしも全ての患者さんに効果が認められるわけではなく、有効性を予測する方法の確立が求められてきました。本研究において、がん細胞が遺伝子変異によって免疫細胞の攻撃から逃れて進展していく様子を表した新規数理モデルを構築し、解析を行いました。がん細胞に蓄積する遺伝子変異には、免疫細胞を活性化させ、がん細胞への攻撃を強くする効果と免疫細胞の攻撃から逃れる効果の両面があることが知られており、その複合的な効果を推測することは困難でしたが、数理モデルを用いることで、初めは免疫活性化の効果によってがん細胞の増殖が鈍り、その後免疫逃避の効果によって増殖速度が上昇するということがわかりました。また、免疫・ゲノム解析によって4つに分類された群のなかで、リンパ球浸潤が多い群と少ない群で、がんが見つかるまでの増殖の仕方が異なることを見出しました。これらの結果をまとめて、大腸がんが免疫の攻撃から逃れる様式が複数あり、それぞれで増殖曲線が異なることを示すことができました。本研究は主に国立がん研究センター研究所のグループと共同で実施しました。本研究は日本経済新聞電子版に掲載されました(https://www.nikkei.com/article/DGXLRSP619870_Z11C21A0000000/)。
脳腫瘍進展の数理モデル解析と最適な治療法の解明 (Aoki et al. 2021 Cancer Res; 日本経済新聞電子版に掲載)
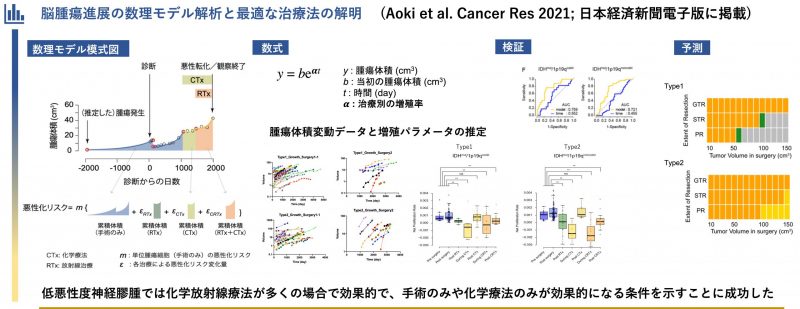
臨床データに基づいた数理モデル解析によって、低悪性度脳腫瘍の悪性化を抑えるのに最適な治療法を明らかにしました。低悪性度脳腫瘍の治療法には、手術、化学療法、放射線治療がありますが、化学療法と放射線治療は遺伝子変異を誘発することで腫瘍の悪性化を促進する可能性も報告されていました。そのため、どの治療をどのタイミングで行うのが悪性化を防ぐために最適な治療であるかはわかっていませんでした。本研究では、日本の10施設にて治療を受けた276名の低悪性度脳腫瘍患者さんの経過中の全てのMRIデータと治療歴を用いて、腫瘍進行に関する数理モデルを構築し解析を行いました。その結果、化学療法や放射線療法などの術後治療は腫瘍の成長を抑制するものの、細胞あたりの悪性化のリスクを増加させることがわかりました。更に、悪性化を防ぐ理想的な治療法が症例毎に異なることを初めて示すことができました。本研究成果は、 “それぞれの患者さんとって”最適な治療戦略を明らかにした、とても重要な知見です。本研究は主に名古屋大学医学部脳神経外科のグループと共同で実施しました。本研究は日本経済新聞電子版 (https://www.nikkei.com/article/DGXLRSP615489_Z20C21A7000000/) などに掲載されました。
大腸癌の新たな転移メカニズムの解明 (Mizukoshi et al. 2020. Int J Cancer. がんプラスに掲載)
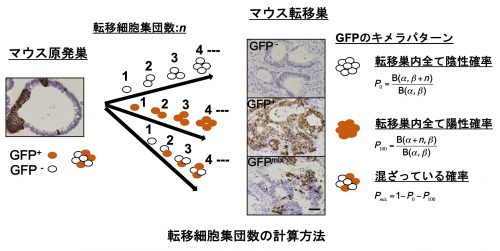
大腸がんの転移が1細胞ではなく細胞集団によって起こることを実験データに基づいた理論計算によって示しました。従来の仮説では、がん細胞が何らかの刺激により間葉系の性質を獲得することでシート状に存在した大腸癌細胞が単一細胞化してシートから離れて周辺組織へ浸潤し、他の臓器への転移が起こると考えられていました。本研究では、40人の大腸がんの患者さんから採取した腫瘍片をランダムに染めてマウスに移植し、その後、転移腫瘍において色のついた細胞の割合に関するデータを取得し、そのデータを最も説明できる転移細胞集団数を推定しました。その結果、肝臓へは2〜3細胞、肺へは3〜35細胞の塊となって転移を起こすことがわかりました。本研究は主に順天堂大学医学研究科分子病理病態学のグループと共同で実施しました。本研究はがんプラス(https://cancer.qlife.jp/news/article11468.html)に掲載されました。
数理モデル解析による膵臓がんの転移パターンの予測(Yamamoto et al. 2017. Cancer Res.)
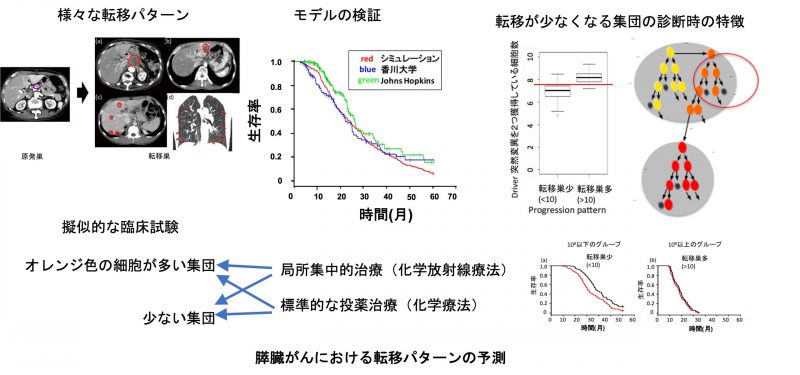
臨床データに基づいた数理モデル解析によって、腫瘍の大きさと特定の遺伝子変異頻度によって、転移が多く起こりやすい膵臓がん患者さんを予測することに成功しました。膵臓がんは5年生存率が10%未満の難治性のがんとして知られ、特に転移による再発が問題となっています。本研究では、先行研究(Haeno et al. 2012 Cell)による膵臓がん細胞の増殖パラメータ、変異パラメータを用いて、膵臓がんの診断時の腫瘍サイズと遺伝子変異頻度のデータから転移が起こる確率や転移巣の数の期待値を予測しました。その結果、予測される転移巣の数が少ない場合は、局所集中的な治療である化学放射線療法をした方が良いことを示しました。臨床データは香川大学とJohns Hopkins病院から提供を受けました。
膵臓がん進展の数理モデル構築と転移予測(Haeno et al. 2012 Cell)
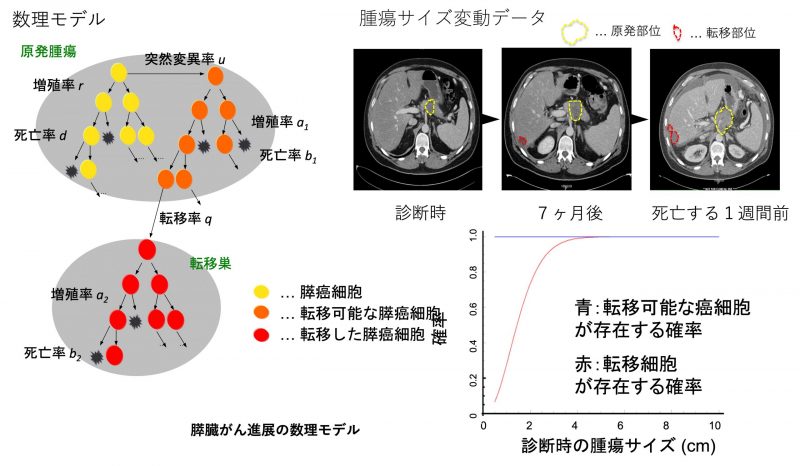
膵臓がんがある大きさで診断された時、転移が起こっている確率と転移細胞集団の数の期待値に関して、確率過程の1つである分枝過程の解析から理論式を導き、臨床データによるモデルパラメータの推定を行い、膵臓がん進展過程を明らかにしました。利用した臨床データは、断層撮影による画像から得られる腫瘍直径データと、剖検時に観察された転移細胞数と転移巣の数に関するデータで、Johns Hopkins病院の共同研究者から101例の提供を受けました。推定したモデルパラメータを用いて、推定に使用したデータとは独立した127例の生存率の予測を行ったところ、数理モデルによる予測は非常によく合うことが確かめられ、一般性が実証されました。さらに、推定されたパラメータを用いると、腫瘍サイズが4cm以上の場合、ほぼ100%転移が存在していることが示唆されました。この研究は膵臓がんの進展過程を臨床データに基づいた数理モデルで表した世界で初めての研究です。本研究は2014年3月18日(火)の日本経済新聞で「がん進行 数式で予測」という記事で紹介されました (https://www.nikkei.com/article/DGXNASGG16008_X10C14A3TJM000/)。
実績
論文
- Kawazu M, Ueno T, Saeki K, Sax N, Togashi Y, Kaneseki T, Chida K, Kishigami F, Sato K, Kojima S, Otsuka M, Kawazoe A, Nishinakamura H, Maeda Y, Yamamoto Y, Yamashita K, Inoue S, Tanegashima T, Matsubara D, Tane K, Tanaka Y, Iinuma H, Hashiguchi Y, Hazama S, Khor SS, Tokunaga K, Tsuboi M, Niki T, Eto M, Shitara K, Torigoe T, Ishihara S, Aburatani H, Haeno H, Nishikawa H, Mano H. HLA Class I analysis provides insight into the genetic and epigenetic background of immune evasion in colorectal cancer with high microsatellite instability. Gastroenterology. 2021 S0016-5085(21)03644-1. (doi: 10.1053/j.gastro.2021.10.010.)
日経電子版(https://www.nikkei.com/article/DGXLRSP619870_Z11C21A0000000/) - Shimizu D, Taniue K, Matsui Y, Haeno H, Araki H, Miura F, Fukunaga M, Shiraishi K, Miyamoto Y, Tsukamoto S, Komine A, Kobayashi Y, Kitagawa A, Yoshikawa Y, Sato K, Saito T, Ito S, Masuda T, Niida A, Suzuki M, Baba H, Ito T, Akimitsu N, Kodera Y, Mimori K. Pan-cancer Methylome Analysis for Cancer Diagnosis and Classification of Cancer Cell of Origin. Cancer Gene Ther. Accepted.
- Takaki M, Haeno H. Mathematical modeling of locoregional recurrence caused by premalignant lesions formed before initial treatment. Front Oncol. Accepted.
- Aoki K, Suzuki H, Yamamoto T, Yamamoto KN, Maeda S, Okuno Y, Ranjit M, Motomura K, Ohka F, Tanahashi K, Hirano M, Nishikawa T, Shimizu H, Kitano Y, Yamaguchi J, Yamazaki S, Nakamura H, Takahashi M, Narita Y, Nakada M, Deguchi S, Mizoguchi M, Momii Y, Muragaki Y, Abe T, Akimoto J, Wakabayashi T, Saito R, Ogawa S, Haeno H, Natsume A. Mathematical modeling and mutational analysis reveal optimal therapy to prevent malignant transformation in grade II IDH-mutant gliomas. Cancer Res. Online ahead of print. 2021. (doi: 10.1158/0008-5472.CAN-21-0985.)
日経電子版(https://www.nikkei.com/article/DGXLRSP615489_Z20C21A7000000/) - Iwai K, Nambu T, Kashima Y, Yu J, Eng K, Miyamoto K, Kakoi K, Gotou M, Takeuchi T, Kogame A, Sappal J, Murai S, Haeno H, Kageyama SI, Kurasawa O, Niu H, Kannan K, Ohashi A. A CDC7 inhibitor sensitizes DNA-damaging chemotherapies by suppressing homologous recombination repair to delay DNA damage recovery. Sci Adv. 7: eabf0197. 2021. (doi: 10.1126/sciadv.abf0197.)
- Sakai K, Yamada Y, Yoshida K, Yoshinaga S, Sato K, Ogata H, Iwasaki T, Kudo S, Asada Y, Kawaguchi I, Haeno H, Sasaki M. Conclusions and suggestions on low-dose and low-dose rate radiation risk estimation methodology. J Radiat Prot Res. 46: 14-23. 2021. (doi: 10.14407/JRPR.2020.00262).
- Fujimoto Y, Morita TY, Ohashi A, Haeno H, Hakozaki Y, Fujii M, Kashima Y, Kobayashi SS, Mukohara T. Combination treatment with a PI3K/Akt/mTOR pathway inhibitor overcomes resistance to anti-HER2 therapy in PIK3CA-mutant HER2-positive breast cancer cells.Sci Rep. 10: 21762. 2020. (doi: 10.1038/s41598-020-78646-y.)
- Hirahara N, Nakamura HM, Sasaki S, Matsushita A, Ohba K, Kuroda G, Sakai Y, Shinkai S, Haeno H, Nishio T, Yoshida S, Oki Y, Suda T. Liganded T3 receptor β2 inhibits the positive feedback autoregulation of the gene for GATA2, a transcription factor critical for thyrotropin production. PLoS One 15: e02276462020. 2020. (doi: 10.1371/journal.pone.0227646.)
- Mizukoshi K, Okazawa Y, Haeno H, Koyama Y, Sulidan K, Komiyama H, Saeki H, Ohtsuji N, Ito Y, Kojima Y, Goto M, Habu S, Hino O, Sakamoto K, Orimo A. Metastatic seeding of human colon cancer cell clusters expressing the hybrid epithelial/mesenchymal state. Int J Cancer. 146:2547-2562. 2020. (doi: 10.1002/ijc.32672.)
がんプラス(https://cancer.qlife.jp/news/article11468.html) - Mimaki S, Watanabe M, Kinoshita M, Yamashita R, Haeno H, Takemura S, Tanaka S, Marubashi S, Totsuka Y, Shibata T, Nakagama H, Ochiai A, Nakamori S, Kubo S, Tsuchihara K. Multifocal origin of occupational cholangiocarcinoma revealed by comparison of multilesion mutational profiles. Carcinogenesis 41: 368-376. 2020. (doi: 10.1093/carcin/bgz120.)
- Yamamoto KN, Nakamura A, Liu LL, Stein S, Tramontano AC, Kartoun U, Shimizu T, Inoue Y, Asakuma M, Haeno H, Kong CY, Uchiyama K, Gonen M, Hur C, Michor F. Computational modeling of pancreatic cancer patients receiving FOLFIRINOX and gemcitabine-based therapies identifies optimum intervention strategies. PLoS One 14: e0215409. 2019. (doi: 10.1371/journal.pone.0215409.)
- Yamamoto KN, Liu LL, Nakamura A, Haeno H, Michor F. Stochastic Evolution of Pancreatic Cancer Metastases During Logistic Clonal Expansion. JCO Clin Cancer Inform. 3:1-11. 2019. (doi: 10.1200/CCI.18.00079.)
- Stein S, Zhao R, Haeno H, Vivanco I, Michor F. Mathematical modeling identifies optimum lapatinib dosing schedules for the treatment of glioblastoma patients. PLoS Comput Biol. 14: e1005924 2018. (doi: 10.1371/journal.pcbi.1005924.)
- Yamamoto KN, Yachida S, Nakamura A, Niida A, Oshima M, De S, Rosati LM, Herman JM, Iacobuzio-Donahue CA, Haeno H. Personalized Management of Pancreatic Ductal Adenocarcinoma Patients through Computational Modeling. Cancer Res. 77:3325-3335 2017. (doi: 10.1158/0008-5472.CAN-16-1208.)
- Yamamoto KN, Ishii M, Inoue Y, Hirokawa F, MacArthur BD, Nakamura A, Haeno H, Uchiyama K. Prediction of postoperative liver regeneration from clinical information using a data-led mathematical model. Sci Rep. 6:34214 2016. (doi: 10.1038/srep34214.)
- Uchi R, Takahashi Y, Niida A, Shimamura T, Hirata H, Sugimachi K, Sawada G, Iwaya T, Kurashige J, Shinden Y, Iguchi T, Eguchi H, Chiba K, Shiraishi Y, Nagae G, Yoshida K, Nagata Y, Haeno H, Yamamoto H, Ishii H, Doki Y, Iinuma H, Sasaki S, Nagayama S, Yamada K, Yachida S, Kato M, Shibata T, Oki E, Saeki H, Shirabe K, Oda Y, Maehara Y, Komune S, Mori M, Suzuki Y, Yamamoto K, Aburatani H, Ogawa S, Miyano S, Mimori K. Integrated Multiregional analysis proposing a new model of colorectal cancer evolution. PLoS Genet. 12:e1005778, 2016. (doi: 10.1371/journal.pgen.1005778.)
- Yamamoto KN, Nakamura A, Haeno H. The evolution of tumor metastasis during clonal expansion with alterations in metastasis driver genes. Sci Rep. 5:15886, 2015. (doi: 10.1038/srep15886.)
- Kobayashi H, Kobayashi CI, Nakamura-Ishizu A, Karigane D, Haeno H, Yamamoto KN, Sato T, Ohteki T, Hayakawa Y, Barber GN, Kurokawa M, Suda T, Takubo K. Bacterial c-di-GMP affects hematopoietic stem/progenitors and their niches through STING. Cell Rep. 11:71-84. 2015. (doi: 10.1016/j.celrep.2015.02.066.)
- Yamamoto KN, Hirota K, Takeda S, Haeno H. Evolution of pre-existing versus acquired resistance to platinum drugs and PARP inhibitors in BRCA-associated cancers. PLoS One. 9:e105724, 2014. (doi: 10.1371/journal.pone.0105724.)
- Haeno H, Maruvka YE, Iwasa Y, Michor F. Stochastic Tunneling of Two Mutations in a Population of Cancer Cells. PLoS One. 8:e65724, 2013. (doi: 10.1371/journal.pone.0065724.)
- Iwami S*, Haeno H*, Michor F. (*co-first authors) A race between tumor immunoescape and genome maintenance selects for optimum levels of (epi)genetic instability. PLoS Comput Biol 8(2):e1002370. 2012. (doi: 10.1371/journal.pcbi.1002370.)
- Haeno H*, Gonen M*, Davis MB, Herman JM, Iacobuzio-Donahue CA, Michor F. (*co-first authors) Computational modeling of pancreatic cancer reveals kinetics of metastasis suggesting optimum treatment strategies. Cell 148(1-2):362-75. 2012. (doi: 10.1016/j.cell.2011.11.060.)
- Hambardzumyan D*, Cheng YK*, Haeno H*, Holland EC, Michor F. (*co-first authors) The Probable Cell of Origin of NF1-and PDGF-Driven Glioblastomas. PLoS One 6(9):e24454. 2011. (doi: 10.1371/journal.pone.0024454.)
- Klinakis A, Lobry C, Abdel-Wahab O, Oh P, Haeno H, Buonamici S, van De Walle I, Cathelin S, Trimarchi T, Araldi E, Liu C, Ibrahim S, Beran M, Zavadil J, Efstratiadis A, Taghon T, Michor F, Levine RL, Aifantis I. A novel tumour-suppressor function for the Notch pathway in myeloid leukaemia. Nature 473, 230-3. 2011. (doi: 10.1038/nature09999.)
- Haeno H, Michor F. The evolution of tumor metastases during clonal expansion. J Theor Biol 263, 30-44. 2009. (doi: 10.1016/j.jtbi.2009.11.005.)
- Haeno H, Levine RL, Gilliland DG, Michor F. A progenitor cell origin of myeloid malignancies. Proc Natl Acad Sci USA 106, 16616-16621. 2009. (doi: 10.1073/pnas.0908107106.)
- Haeno H, Iwasa Y, Michor F. The evolution of two mutations during clonal expansion. Genetics 177, 2209-2221. 2007. (PMID: 18073428)
- Haeno H, Iwasa Y. Probability of resistance evolution for exponentially growing virus in the host. J Theor Biol 246, 323-331. 2007. (PMID: 17306832)
日本語総説
- 巌佐庸、 波江野洋「進化プロセスとしての発がん」 実験医学増刊号2017年3月 Vol.35 No.5, p227-230
- 波江野 洋、山本 君代 「がん進展過程における薬剤耐性獲得の数理モデル」2014年9月 がん分子標的治療 Vol. 12, No. 3, p324-p329
- 波江野 洋、山本 君代 「がんにおける突然変異蓄積過程の数理モデル」2014年7月 実験医学増刊号 Vol. 32, No. 12, p208-p212
- 波江野 洋 「突然変異の蓄積を含むがんの動態の数理モデリング」2013年10月 実験医学 Vol. 31, No. 18, p2939-p2944
- 岩見 真吾、佐藤 佳、合原 一幸、波江野 洋、錦織 桃子、高折 晃史、小柳 義夫 「細胞のダイナミクスを記述する──疾患研究のイノベーションへ」2013年10月 科学 Vol. 83, p1184-p1189

