櫻井 雅之さくらい まさゆき
ゲノム&RNA 編集病態学
研究テーマ
- DNA及びRNA塩基の化学構造修飾編集機構が担う生命現象
- RNAが調節するゲノム安定性維持及び遺伝子発現制御機構
- これら制御機構の破綻により引き起こされる疾患の発症分子機構 -DNA損傷/修復異常/変異 -DNA:RNA間の異常構造体形成による細胞ストレス -細胞のがん化/炎症惹起/細胞死/細胞老化
- 分子レベルからの解析に基づいたこれら疾患原因の検出と診断/治療技術の開発
2001年東京大学工学部化学生命工学科卒(渡辺公綱教授)、2006年東京大学大学院新領域創成科学研究科先端生命科学専攻修了、博士(生命科学)取得(鈴木勉教授)。同年JBIC機能性RNAプロジェクト研究員。2010年より学振海外特別研究員、米国フィラデルフィアのWistar Instituteへ (Prof. Kazuko Nishikura)。同研究所Staff Scientist, Senior Staff Scientist, Research Assistant Professorを経て2018年より東京理科大学生命医科学研究所、分子病態学研究部門講師。
「生命のセントラルドグマ」とはDNAから適宜必要な遺伝子をRNAへと転写し、機能発現体であるタンパク質を産生する遺伝子発現の流れである。各段階において多彩な発現調節機構が働くことにより、適切な遺伝子発現が保たれているが、当研究室は遺伝子情報そのものであるDNAとRNAを構成する[A,G,C,T(U)]4種の塩基構造を修飾編集する機構を主題としている。そして機能性構造物としてのDNA及びRNAとタンパク質の認識機構において塩基修飾が与える影響を試験管内、細胞内、組織/個体内の各段階での解析を行い、分子レベルの機構解明から生物学的な意義までを解明することを目指している。また、これら機構の破綻により引き起こされる細胞障害や疾患の分子病態の解明を進めている。そのために生体分子として修飾を含めて核酸やタンパク質を合成や精製から反応解析までを駆使し、さらにこれら分子が細胞に与える影響を、遺伝子制御やエピゲノム・転写・翻訳機構の解析、細胞周期や細胞死・炎症・がん化などの解析を行っている。特に、一つの実験から得られる知見を多角的に捉えつつ、複数のアプローチから一つの現象を解析し、既成概念にとらわれることなく新発見へ挑戦することを研究室のモットーとしている。
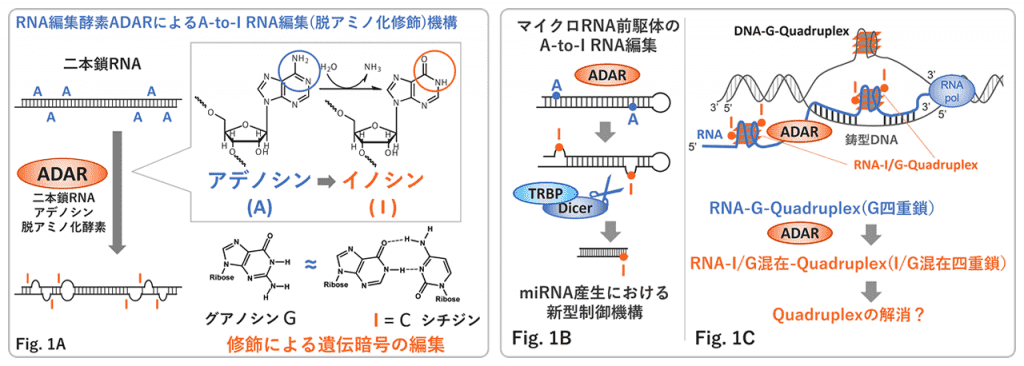
RNA鎖構造にRNA編集が与える影響とその生物学的意義の解明
アデノシン(A)をイノシン(I)へと変換する機構は、RNA編集と呼ばれる。RNA編集では通常、編集酵素であるADAR(Adenosine Deaminase Acting on dsRNA)により、二本鎖RNA領域内のAが脱アミノ化されIへと修飾される(Fig. 1A)。本アプローチでは、RNA編集が二本鎖RNAに起きた場合、各対象RNAの構造に与える影響を分子レベルで解析し、その変化が与えるRNAの機能、そして最終的な生物学的意義を探求する。具体的には、マイクロRNA前駆体におけるRNA編集が与える新しい型の制御機構(Fig. 1B)と、グアノシン(G)に富むRNA鎖が形成しやすく、RNAの機能発現の際に障害となり細胞ストレスの原因となるグアノシン4重鎖(G-quadruplex) (Fig. 1C)に対するRNA編集の効果とその作用分子機序を試験管内、細胞内、組織/個体内において解明する。
RNAのDNAとのハイブリッド鎖形成機構におけるRNA編集の機能解析
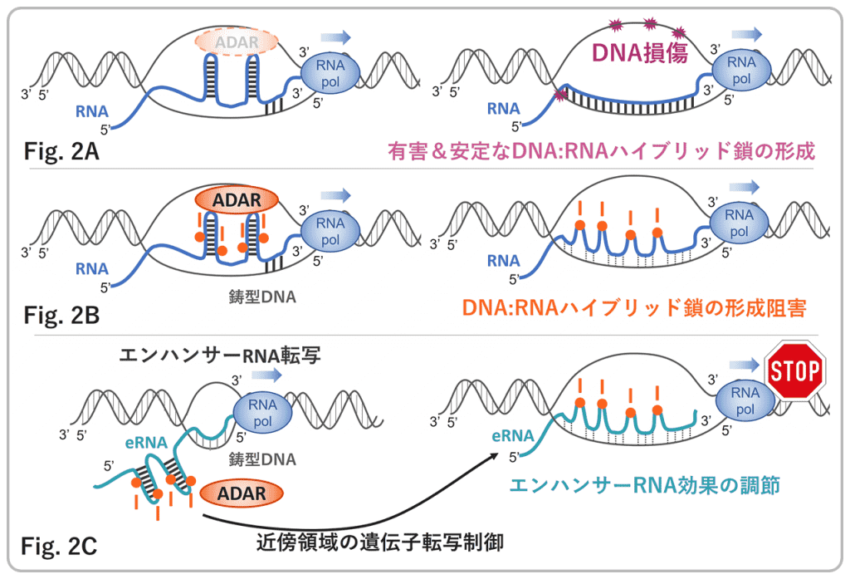
通常、転写によって新規合成されたRNA鎖は鋳型DNAから即座に解離するが、一部の配列ではRNA鎖が再度鋳型DNAと対合して二本鎖DNAよりも安定なRNA:DNAハイブリッド鎖を形成したまま留まり、DNAセンス鎖は一本鎖のままとなる (Fig. 2A)。この構造はR-loopと呼ばれ、ゲノム構造の不安定化や転写産物のプロセシング阻害を引き起こすため新たな内因性の危険因子として注目されている。ヒトでは運動失調動眼失行症2型(AOA2)・脆弱性X症候群(FXS) ・前頭側頭型認知症(FTD)・筋萎縮性側索硬化症(ALS)などの重大難治疾患の原因となることが報告されている。我々はこのRループ構造解消因子遺伝子群の同定を進めており、その中にRNA編集酵素群を見出している(Fig. 2B)。一方、非コードRNAのうち、その発現が近傍の各遺伝子の発現を促進する性質をもつ一群がエンハンサーRNA(eRNA)として発見されているが、その分子作用機序は不明である。我々はこのeRNAが対象遺伝子のプロモーター領域でRNA:DNAハイブリッド鎖を形成し、DNAを開いた構造に維持することでその転写を促進していると仮説を立てている(Fig. 2C)。本アプローチでは、RNA:DNAハイブリッド鎖の形成及び解消におけるRNA編集酵素と補因子遺伝子群の作用機序の解明から、その生物学的意義にせまる。
RNA編集酵素の変異による炎症惹起機構と編集機構破綻によるDNA損傷と細胞周期異常
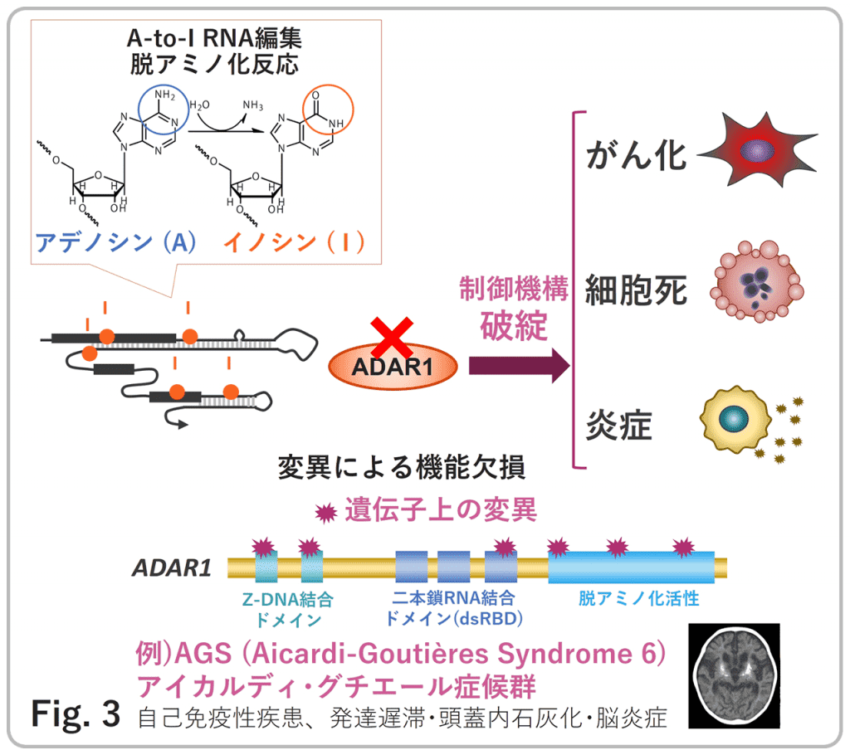
ADARの機能の一つとして、細胞内におけるインターフェロン(IFN)抑制効果が知られている。さらに、上述のAGS疾患の原因遺伝子の一つにADAR1が報告されている(Fig. 3)。しかしながらその発症分子機構には未解明な点が多いままである。本アプローチではAGS疾患患者にみられるADAR1遺伝子上の変異導入体を用いて、そのIFN惹起経路を解明する。惹起分子の候補としては二本鎖RNA構造またはRNA:DNAハイブリッド鎖が考えられ、RNA編集酵素による制御機構の検証を進める。ADARの発現抑制が引き起こすもう一つの顕著な現象として過剰なDNA損傷の蓄積と細胞周期及び損傷チェックポイントの不具合に起因すると考えられる分裂期細胞周期停止による細胞死:Mitotic Catastropheを我々は発見している。本アプローチではADARが与えるがん細胞と正常細胞への影響の差異の探求から、RNA編集が制御するDNA安定性及び修復機構と細胞命運決定経路の同定を進める。
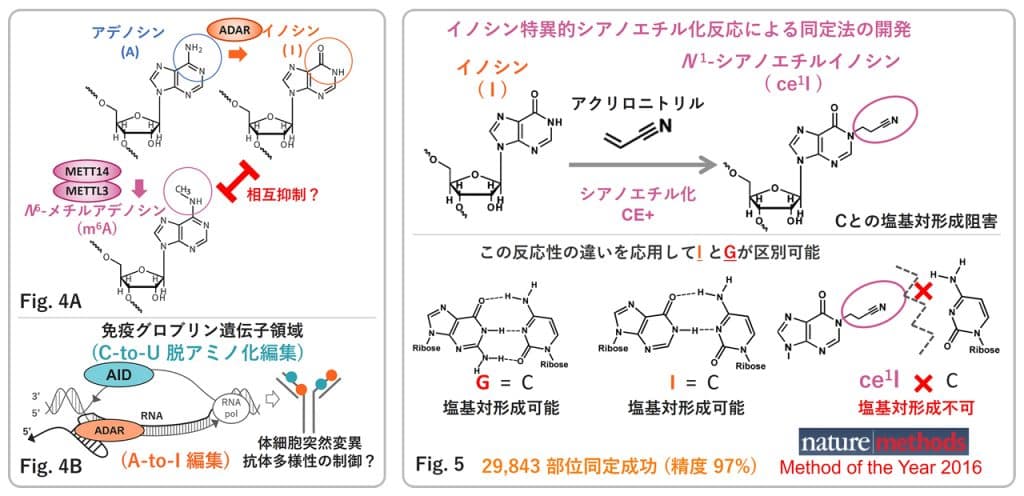
多様な塩基修飾間の相互作用と新規RNA修飾/構造体高精度検出法の開発
ADARによるA-to-I RNA編集機構以外にも、RNAまたはDNAにおける塩基修飾はmRNA及び機能未知なノンコーディングRNAでも見つかっている。近年mRNAにおいて発見され、機能研究が盛んであるN6-メチルアデノシン(m6A)は、その修飾によりA-to-I RNA編集機構に対して阻害的な作用をもつと考えられる(Fig. 4A)。一方、免疫系B細胞では抗原受容体産生過程において、クラススイッチ及び体細胞突然変異を起こす際、AID(Activation induced cytidine deaminase)と呼ばれるDNA/RNAのシチジン(C)脱アミノ化編集酵素が必須であることが知られている。本アプローチでは、A-to-I RNA編集機構とこれら他の塩基編集修飾機構との相互作用の分子機構を明らかにし、その生物学的意義を探求する(Fig. 4B)。しかし上述してきたRNA構造体及びRNA:DNAハイブリッド鎖構造、塩基修飾の検出技術は未だ発展段階にあり、正確な分子機構の解明には検出技術の発展が必要である。将来的には核酸構造や塩基修飾が関与する生命現象及び疾患の関係解明により、得られる知見を活用することで核酸構造や塩基修飾の高精度検出による病態の診断と治療法の開発への応用が可能となる。私自身イノシンの検出技術として人工化学修飾導入法を応用した検出技術開発の経験を持ち(櫻井ら、2014&2010)Nature Methods誌のMethod of the Year 2016に選出されている(Fig. 5)。この経験を生かし、現在m6Aなど他の塩基修飾の新規検出法の開発を進めている。
代表的な業績
- Sakurai, M., Shiromoto, Y., Ota, H., Song, C., Kossenkov, A.V., Wickramasinghe, J., Showe, L.C., Skordalakes, E., Tao, H.Y., Speicher, D.W., and Nishikura, K. “ADAR1 controls apoptosis of stressed cells by inhibiting Staufen-mediated mRNA decay.” Nat. Struct. Mol. Biol. 24: 534-543 [DOI: 10.1038/nsmb.3403, PMID: 28436945] (2017).
- Gumireddy, K., Li, A., Kossenkov, A.V., Sakurai, M., Yan, J., Li, Y., Xu, H., Wang, J., Zhang, PJ., Zhang, L., Showe, L., Nishikura, K., and Huang, Q. “The mRNA-edited form of GABRA3 suppresses GABRA3-mediated Akt activation and breast cancer metastasis.” Nat. Commun. 7: 10715 [DOI: 10.1038/ncomms10715, PMID: 26869349] (2016).
- Okada, S., Sakurai, M., Ueda, H., and Suzuki, T. “Biochemical and Transcriptome-Wide Identification of A-to-I RNA Editing Sites by ICE-Seq” Methods Enzymol. 560: 331-35 [DOI: 10.1016/bs.mie.2015.03.014, PMID: 26253977] (2015).
- Suzuki, T., Ueda, H., Okada, S., and Sakurai, M. “Transcriptome-wide identification of adenosine-to-inosine editing using the ICE-seq method.” Nat. Protoc. 10: 715-732 [DOI: 10.1038/nprot.2015.037, PMID: 25855956] (2015).
- Sakurai, M., Ueda, H., Yano, T., Okada, S., Terajima, H., Mitsuyama T., Toyoda, A., Fujiyama, A., Kawabata, H., and Suzuki, T. “A biochemical landscape of A-to-I RNA editing in the human transcriptome.” Genome Res. 24: 522-34 [DOI: 10.1101/gr.162537.113, PMID: 24407955] (2014). *This work is awarded for the Nature Method “METHOD OF THE YEAR 2016”: Li X., Xiog, X., and Yu C. “Epitranscriptome sequencing technologies: decoding RNA modifications.” Nature Methods 14: 23-31 (2017).
- Ota, H*., Sakurai, M.* (*Equal contribution as a first author), and Gupta, R., Valente, L., Wulff, B.E., Ariyoshi, K., Iizasa, H., Davuluri, R.V., and Nishikura, K. “ADAR1 Forms a Complex with Dicer to Promote MicroRNA Processing and RNA-Induced Gene Silencing.” Cell 153: 575-589 [DOI: 10.1016/j.cell.2013.03.024, PMID: 23622242] (2013).
- Sakurai, M., Yano, T., and Suzuki, T. “Biochemical identification of A-to-I RNA editing sites by the Inosine Chemical Erasing (ICE) method.” Methods Mol. Biol. 718: 89-99 [DOI: 10.1007/978-1-61779-018-8_5, PMID: 21370043] (2011).
- Sakurai, M., Yano, T., Kawabata, H., Ueda, H., and Suzuki, T. “Inosine cyanoethylation identifies A-to-I RNA editing sites in the human transcriptome.” Nat. Chem. Biol. 6: 733-740 [DOI: 10.1038/nchembio.434, PMID: 20835228] (2010).
- Sakurai, M., Watanabe, Y., Watanabe, K., and Ohtsuki, T. “A protein extension to shorten RNA: Elongated elongation factor Tu recognizes the D-arm of T-armless tRNAs in nematode mitochondria.” Biochem J. 399: 249-56 [DOI: 10.1042/BJ20060781, PMID: 16859488] (2006).
- Sakurai, M., Ohtsuki, T., and Watanabe, K. “Modification at position 9 with 1-methyladenosine is crucial for structure and function of nematode mitochondrial tRNAs lacking the entire T arm.” Nucleic Acids Res. 33: 1653-1661 [DOI: 10.1093/nar/gki309, PMID: 15781491] (2005).
- Sakurai, M., Ohtsuki, T., Suzuki, T., and Watanabe, K. “Unusual usage of wobble modifications in mitochondrial tRNAs of the nematode Ascaris suum.” FEBS Lett. 579: 2767-2772 [DOI: 10.1016/j.febslet.2005.04.009, PMID: 15907479] (2005).
- Suematsu, T., Sato, A., Sakurai, M., Watanabe, K., and Ohtsuki, T. “A unique tRNA recognition mechanism of Caenorhabditis elegans mitochondrial EF-Tu2.” Nucleic Acids Res. 33: 4683-4691 [DOI: 10.1093/nar/gki784, PMID: 16113240] (2005).
- Ohtsuki, T., Sakurai, M., Sato, A., and Watanabe, K. “Characterization of the interaction between the nucleotide exchange factor EF-Ts from nematode mitochondria and elongation factor Tu.” Nucleic Acids Res. 30: 5444-5451 [PMID: 12490713] (2002).
- Sakurai, M., Ohtsuki, T., Watanabe, Y., and Watanabe, K. “Requirement of modified residue m1A9 for EF-Tu binding to nematode mitochondrial tRNA lacking the T arm.” Nucleic Acids Res.Suppl. 1: 237-238 [DOI: 10.1093/nar/gki309, PMID: 15781491] (2001).
- Song, C., Sakurai, M., Shiromoto, Y., Ota, H., Nishikura, K. “Functions of the RNA Editing Enzyme ADAR1 and Their Relevance to Human Diseases.” Gene 7: 129 [DOI: 10.3390/genes7120129, PMID: 27999332] (2016).
- Nishikura, K., Sakurai, M., Ariyoshi, K., and Ohta, H. “Antagonistic and stimulative roles of ADAR in RNA silencing An editor’s point-of-view.” RNA biology 10: 1240-1247 [DOI: 10.4161/rna.25947, PMID: 23949595] (2013).
- Wulff, B.E., Sakurai, M., and Nishikura, K. “Elucidating the inosinome: Global approaches to adenosine-to-inosine RNA editing.” Nat. Rev. Genet. 12: 81-85 [DOI: 10.1038/nrg2915, PMID: 21173775] (2011).
- 櫻井雅之, 太田博允, 西倉和子 「RNA編集酵素ADAR1のRNAi促進機能」実験医学 31: 2615-2618 (2013).
- 櫻井雅之, 鈴木勉 「転写後修飾によるRNA機能制御」細胞工学 28: 149-155 (2009).
- Sakurai, M., and Suzuki, T., “Method for detection of inosine-containing site in RNA”. WO2007018169A1, 7 Aug 2006, (International application). 櫻井雅之, 鈴木勉. 特開2008-259425 「RNA中のイノシン化部位の検出方法」2008年10月30日公開, 特願2005-229335, 出願日2005年8月8日.

